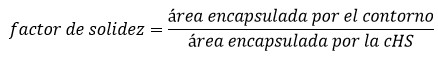INTRODUCCIÓN
En el mundo contemporáneo los laboratorios de microbiología juegan papeles importantes para la sociedad, en aplicaciones tan diversas que pueden ir desde la detección de enfermedades hasta el monitoreo ambiental.(1,2) Uno de los procedimientos habituales dentro de estos laboratorios es la detección y conteo de microorganismos o de sus unidades formadoras de colonia (UFC), en diferentes sustancias, ya sea para analizar los microorganismos en sí o para el estudio de las propias sustancias.(3)
Actualmente se encuentran disponibles en el mercado diferentes equipamientos para realizar dichas labores de conteo de manera automática o semiautomática, pero la inversión necesaria para su adquisición suele ser elevada,(4,5) con una amortización que puede ser considerada relativamente lenta. Esta situación provoca que en muchos laboratorios aún estos procesos se hagan de forma “tradicional” mediante la observación de las muestras en los microscopios por parte de expertos. Este método de observación mediante microscopios requiere de gran cantidad de tiempo y esfuerzo por parte del personal del laboratorio, puede producir fatiga ocular en los observadores y ser diferente el conteo realizado entre diferentes especialistas,(6,7) sobre todo cuando el número de microorganismos involucrados en el proceso de conteo es alto.(8)
Tratando de evitar estos inconvenientes, en los últimos años se han desarrollado varios métodos y software para el conteo de microorganismos, de los cuales una gran parte se basa en el uso de imágenes digitales de las muestras a analizar. Quizás la herramienta de análisis y procesamiento de imágenes más expandida en este contexto ha sido el software ImageJ, o su nueva versión Fij, pero aun así la exactitud de los resultados de estas herramientas varía en dependencia del contexto donde sean aplicados, por lo que hasta el presente ha sido difícil contar con métodos generales para realizar el conteo de microorganismos en imágenes digitales.
La imposibilidad de generalizar los procedimientos y métodos en el conteo de microorganismos en imágenes digitales se debe a la amplia cantidad de características y circunstancias que se pueden encontrar en los mismos y en las muestras analizadas, tales como la forma, el tamaño, textura, solapamiento entre ellos, escala a la que son tomadas las imágenes, contraste entre el fondo y los microorganismos, etc.(9)
Es por ello que la mayoría de los métodos y algoritmos son ajustados en base a experimentos específicos, mediante la configuración de los parámetros de los mismos.(10)
Estos algoritmos de conteo en imágenes digitales se pueden dividir en dos grandes grupos: técnicas de visión por computador (VC) y procesamiento digital de imágenes (PDI), y técnicas de machine learning (ML). Con las técnicas de PDI y VC se puede lograr una detección que depende solo de las características de la imagen analizada, mientras que con las variantes de técnicas de ML se logra aumentar considerablemente la eficiencia en comparación con las técnicas anteriores, pues dichas técnicas pueden obtener modelos que aprenden patrones de las imágenes, pero se requiere para ello grandes cantidades de datos de experimentos.
Dentro de las técnicas de PDI y VC empleadas para realizar estos conteos se encuentra el empleo de métodos de umbralado multinivel,(11) operaciones morfológicas,(12) detección de bordes,(9) algoritmo watershed,(6,13,14)y transformada de distancia,(13) entre otras. En varios trabajos los algoritmos son precedidos por una serie de pasos denominados preprocesamiento, los cuales pueden abarcar la conversión entre espacios de colores, filtrado de las imágenes y el ajuste de contraste.
Observando el comportamiento de las investigaciones en este escenario científico, profesores-investigadores del Departamento de Control Automático de la Universidad Central Marta Abreu de Las Villas (UCLV), unieron sus esfuerzos con especialistas del Instituto de Biotecnología de las Plantas (IBP) de la UCLV para desarrollar un método para el conteo de bacterias y levaduras en bioproductos de origen microbiano obtenidos por fermentación, los cuales se encuentran aún en fase de estudios pero han demostrado aplicaciones útiles en diversas industrias.(15)
Es de importancia mencionar que en la bibliografía consultada no se tiene referencia de hacer la detección y conteo de varios microorganismos de diversas naturalezas, como son bacterias y levaduras, en la misma imagen; pues la mayoría de ellas se enfoca en contar muestras de un solo tipo de microrganismos.
El objetivo de este trabajo es proponer un nuevo método para el conteo de bacterias y levaduras en imágenes digitales, bajo diferentes magnificaciones, tomadas a bioproductos de origen microbiano obtenidos por fermentación.
MÉTODOS
En la realización del presente trabajo se utilizaron muestras de bioproductos de origen microbiano obtenidos por fermentación que se encuentran bajo investigaciones en el Instituto de Biotecnología de las Plantas (IBP), los cuales están formados por un medio de cultivo y diversos microrganismos.
En este caso se seleccionaron muestras donde los organismos vivos que se encuentran en los compuestos fueran bacterias y levaduras. El proceso de preparación de las muestras queda fuera del alcance de este trabajo, el cual comienza a partir del momento en que son adquiridas las imágenes.
Según se tienen referencias por parte de la bibliografía especializada y por la experiencia del personal del laboratorio, en estos bioproductos las bacterias alcanzan un tamaño que oscila entre 1 µm y 3 µm, mientras que las levaduras pueden alcanzar hasta 10 µm. Además, es común que en este tipo de compuestos las bacterias sean mayor cantidad que las levaduras.
El sensor empleado para la toma de imágenes de las muestras fue una cámara digital modelo HDCE-X, con un sensor CMOS de ½", con una resolución de 2592 píxeles por 1944 píxeles (5 Mp), donde el tamaño del pixel es de 3.2µm X 3.2µm, con un tiempo de exposición ajustable. Esta cámara es conectada a través de un cable USB a una computadora donde se almacenan las imágenes en formato JPG con 24 bits de profundidad (8 bits por canal).
Las imágenes fueron adquiridas con la cámara mencionada y un microscopio de la serie Olympus BH. Se emplearon fundamentalmente dos tipos de magnificaciones: magnificación 40x (PL40, 0.65 apertura numérica and 0.17 de distancia de trabajo) obteniéndose un factor de escala de 2.3 pixeles/ μm; y magnificación 100x (HI plan 100/1.25 con inmersión de aceite) siendo el factor de escala de 6.0 pixeles/ μm.
El código del algoritmo fue realizado en lenguaje Python 3, empleando la librería de procesamiento digital de imágenes OpenCV en su versión 4.0.0.
Descripción del método propuesto
El método propuesto para la detección y conteo de bacterias y levaduras en imágenes digitales se compone de varias fases o etapas, como se puede apreciar a continuación:
- Leer la imagen.
- Fase I:
- Conversión de espacios de colores BGR a gris
- Operación morfológica
- Umbralado
- Detección de contornos
- Fase II:
- Análisis estadístico
- Fase III:
- Clasificación
- Brindar conteo
La Fase I tiene como objetivo delimitar los posibles microorganismos en la imagen. En esta fase la imagen es leída como una imagen de 3 canales. (Fig. 1a). Luego la imagen es convertida a escala de grises, y a esta nueva imagen se le aplica un primer proceso de operaciones morfológicas, consistente en el método Black Top-hat con el objetivo de resaltar en la imagen las siluetas de los microorganismos. Dicha operación morfológica consiste en la diferencia entre el proceso de hacer un cierre morfológico sobre la imagen en escala de grises y la propia imagen en escala de grises. Como esta operación morfológica se encuentra compuesta de una operación de cierre sobre la imagen, dicha operación en general tiende a revelar áreas que son más oscuras que sus vecinos, en relación con el tamaño del elemento estructurante.
Para este proceso se empleó un elemento estructurante o strel compuesto de 1 en todas sus posiciones de tamaño de 9 píxeles. La selección del tamaño del strel se ajustó a través de aplicar la misma operación en diferentes experimentos, observando cual era el tamaño más adecuado para cubrir la gama de los experimentos.
Una vez aplicada la operación morfológica a la imagen se procede a aplicar un proceso de umbral sobre la misma, seleccionando el método de Otsu para aplicar un proceso de binarización global, obteniéndose de esta manera una imagen binaria codificada en 0 y 1 (Fig. 1-b).
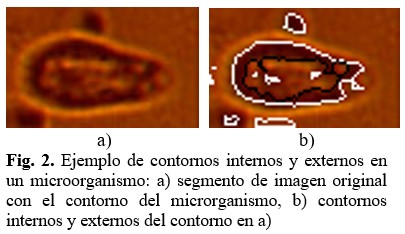
Luego de obtenida la imagen binaria se aplica un proceso para la obtención de contornos presentes en la misma. En este caso solo se consideran válidos para el análisis los contornos externos que provocan las siluetas de los microrganismos, pues en ocasiones los microrganismos suelen presentar ciertos patrones en las imágenes que tienden a ser clasificados como contornos internos, tal como ilustra en la figura 2. En dicha figura los contornos externos son dibujados con línea blanca y los contornos internos con líneas negras. (Fig. 2).
Cuando todos los contornos externos de los microorganismos que aparecen en la imagen han sido determinados, comienza la Fase II, la cual tiene como objetivo determinar cuál es la moda del tamaño de los contornos de las bacterias presentes en la imagen.
Con este fin, a cada contorno externo hallado se le determina el polígono convexo que rodea dicho perímetro (cHS), es decir, aquel polígono de menor área que contiene todos los puntos del contorno.
Como el algoritmo no contempla ningún proceso de filtrado sobre la imagen, en ocasiones aparecen pequeños contornos que pueden ser despreciados por ser demasiados pequeños. Es por ello que se decide calcular el área encapsulada por el polígono determinado, y utilizar un parámetro de ajuste para seleccionar los contornos. En este caso si dicha área es mayor de 1 píxel se toma en cuenta para los siguientes análisis, en caso contrario no se tiene en cuenta en la segunda fase debido a que áreas tan pequeñas pueden pertenecer a ruido en las imágenes o a impurezas en el ambiente donde se encuentran los microorganismos,(9) igual criterio que se sigue en la tercera fase. Este parámetro fue ajustado en base a las observaciones realizadas durante diferentes experimentos.
Si la envoltura convexa encapsula un área que merece ser tenida en consideración, entonces, al conjunto binario de los pixeles internos encapsulados por el contorno analizado se le determina su área, con el objetivo final de calcular el factor de solidez del contorno mediante el empleo de la Ecuación 1.
El objetivo del factor de solidez es determinar cuan regular puede ser un contorno encontrado, el cual se debe corresponder con el criterio de que son los contornos de bacterias o levaduras bien definidas y que no están formando aglomeraciones dado que mientras más convexo mayor será su factor de solidez. Esta idea del factor de comparación ya ha sido manejada en investigaciones anteriores,(13) pero con diferente enfoque.
En este punto del algoritmo el factor de comparación de la solidez de un contorno se fijó en 0.90, considerando así para el análisis estadístico obtener contornos lo más convexos posibles.
Si el contorno analizado cumple con el criterio de solidez entonces se calcula el mínimo círculo que encapsula a dicho contorno (mEC), es decir, el círculo de área mínima que contiene todos los puntos del contorno, tal como se puede observar en la figura 3. En este paso la parte entera de la medida del radio del mEC es tenida en cuenta a la hora de determinar la primera y la segunda moda de los radios de los contornos; en caso contrario no es tenido en cuenta. (Fig. 3).
Es de mencionar que el área que ocupan los píxeles que son encapsulados por los contornos no es usada directamente como criterio de comparación puesto que el agrupamiento de varios microorganismos pueden tener un área similar al área que puede pertenecer a una levadura, que son los microorganismo de mayor tamaño que aparecen en la imagen. Otro aspecto fundamental es que el método es concebido para que funcione bajo diferentes magnificaciones del conjunto microscopio-cámara, por lo cual a diferentes magnificaciones el área que ocupan los microorganismos es diferente.
Una vez concluida la segunda fase de análisis estadístico se da paso a la Fase III que tiene como objetivo clasificar los microorganismos detectados en bacterias y levaduras, para finalmente brindar un conteo de los mismos.
Para realizar dicha tarea se vuelven a analizar los todos los contornos externos determinados anteriormente.
Para cada uno de estos contornos se determina nuevamente la cHS que lo encapsula y el área de la misma, y se emplea el mismo criterio que se empleó en la fase dos para discriminar entre los contornos, siendo los contornos muy pequeños excluidos del proceso de clasificación. Si el contorno llega a tener un área mínima establecida entonces se le calcula el radio del mEC que encapsula al contorno.
En este punto del algoritmo se sitúa un criterio de comparación que permite que los contornos pequeños, aquellos cuyo radio del mEC sea menor que 0.5 píxeles sean desestimados y no se clasifiquen; en caso contrario se prosigue con su análisis. Este criterio de selección fue ajustado en base a los experimentos realizados.
Al cumplir el contorno con este criterio de selección entonces aparece un segundo criterio que permite discernir si el contorno analizado tiene mayor probabilidad de ser una bacteria o no. Dicho criterio de ajuste se basa en comparar el radio del mEC hallado con el mayor valor de las dos modas de los radios determinados anteriormente. Si dicho radio es menor que 3 veces el mayor de estos valores, entonces tiene mayor posibilidad de ser una bacteria; en caso contrario tiene mayor posibilidad de ser una levadura o una unión de varios microorganismos, por lo que estos contornos sufren de otro proceso para una mejor clasificación.
Al cumplir con ambos criterios de selección se calcula de nuevo su factor de solidez y en esta ocasión si es mayor de 0.7 es clasificada como bacteria; en caso contrario se clasifica como unknown dada la irregularidad de la forma.
Los contornos más grandes sufren de otro proceso de clasificación. Estos contornos pueden ser compuestos por levaduras, o por agrupaciones de bacterias y levaduras, es por ello que se precisa de realizar un análisis diferente para su clasificación. En una nueva imagen en negro, estos contornos son dibujados y rellenados, para luego aplicárseles dos procesos de operaciones morfológicas.
El primero de ellos consta de un proceso de erosión con el fin de separar posibles uniones de diversos contornos por si el contorno en estudio se encuentra compuesto por más de un microorganismo, en el cual el tamaño del elemento estructurante a emplear se selecciona de acuerdo a la moda de los radios de los mEC: si dicha moda es menor que dos, el tamaño del elemento estructurante queda determinado en 2 píxeles, y si es mayor que 2 entonces se asume que el tamaño del elemento estructurante queda fijado por la parte entera de la moda de los radios.
Acto seguido se aplica un proceso de dilatación al contorno ya erosionado con el objetivo de aumentar el área de los contornos remanentes del proceso de erosión, pero evitando que estos se vuelvan a unir. Para el proceso de dilatación el tamaño del elemento estructurante a emplear se disminuye en una unidad respecto al tamaño del elemento estructurante que se empleó en el proceso de erosión.
Una vez realizados estos dos procesos se procede a determinar los nuevos contornos externos que aparecen en esta nueva imagen. Para cada uno de estos contornos se calcula el radio del mEC.
En este punto se vuelve a emplear el criterio de comparación utilizado anteriormente: si el radio del mEC es mayor que 0.5 píxeles se procede a su clasificación, en caso contrario se descarta el contorno por ser demasiado pequeño.
Si el radio del nuevo contorno cumple el requisito para ser clasificado, se compara con la parte entera del mayor valor de la primera y segunda moda de los radios de los mEC determinados en la fase estadística. Si este nuevo radio es menor que el mayor valor de las dos modas de los radios, entonces es clasificado ese contorno como una bacteria, en caso contrario es clasificada como levadura.
Finalmente, el conteo es brindado a los usuarios sobre la misma imagen, resaltando en círculos de color verde las bacterias, en círculos color rojo las formas desconocidas y en rectángulo color malva las levaduras.
Flujo de trabajo realizado con el software ImageJ para el conteo de bacterias y levaduras
Como se mencionó anteriormente el software ImageJ constituye una de las herramientas más difundidas en el ámbito científico para el procesamiento y estudio de imágenes digitales. Su gran uso se debe, entre otras muchas prestaciones, a que es un software de licencia abierta por el cual no hay que pagar para su utilización, y aunque inicialmente fue concebido para un uso en campos relacionados con la biología, ha logrado adentrarse en otros campos de las ciencias. Además, recibe soporte activo por la comunidad científica adicionándole nuevas funciones y algoritmos, y posibilita la aceleración del flujo de trabajo mediante la creación de macros y pluggins personalizados, y el procesamiento de imágenes en lotes.
Para el conteo de bacterias y levaduras en las imágenes empleadas en la presente investigación se siguió un flujo de trabajo que constó de los pasos mostrados a continuación:
- Abrir la imagen.
- Aplicar la opción “Subtract Background”:
- Convertir la imagen a escala de grises
- Aplicar la opción “Convert to mask”
- Aplicar la opción "Fill Holes"
- Aplicar la opción "Watershed"
- Aplicar la opción "Analyze Particles..."
Una vez abierta la imagen con el software, se le aplica un algoritmo para sustraer el fondo de la imagen, es decir, la suspensión donde se encuentran ubicados los microrganismos. Para esto a la opción rolling se le asignó un valor de 60. Luego la imagen es convertida a escala de grises, utilizando la opción “8 bit” ofrecida por el software. Esta nueva imagen es transformada a binaria mediante la opción Convert to mask, donde la opción de BlackBackground del software fue habilitada para obtener contornos blancos sobre fondo negro.
A esta imagen binaria se le aplica la opción de rellenar huecos con el fin de que aquellos contornos donde quedaron conjuntos de pixeles interiores de otro color (denominados huecos) sean rellenados. El algoritmo Watershed es aplicado con el fin de separar aquellos contornos donde puedan existir agrupaciones de microorganismos en diferentes regiones. Es de mencionar que, según se pudo observar en la literatura consultada, este algoritmo es uno de los más empleados para la delimitación y separación de contornos de microorganismos en este tipo de aplicaciones.
Por último, una vez delimitados los contornos de los microrganismos, se procede a utilizar la opción Analyze Particles.. del software ImageJ, el cual permite seleccionar qué tipos de partículas usted desea analizar basándose fundamentalmente en dos criterios: su área y cuan redondo es el contorno. En este paso es donde se separaron para análisis los dos microrganismos a analizar, seleccionándose para el caso de las bacterias los contornos cuya área oscilara entre 0 y 50 píxeles cuadrados, mientras que para las levaduras se seleccionaron contornos con un área superior a los 51 píxeles cuadrados.
Como se puede apreciar, el flujo establecido con el software ImageJ para procesar las imágenes con el fin de realizar el conteo de bacterias y levaduras fue el mismo, excepto en el último paso donde la configuración de los parámetros al aplicar el método de Analyze Particles.. es diferente, discriminándose los contornos por su área.
RESULTADOS
Durante el transcurso de esta investigación se realizaron varios experimentos en los cuales la suspensión donde se encontraban los microorganismos, la tintura que se le dio a la suspensión, el grado de magnificación de conjunto microscopio-cámara, el enfoque, entre otras características, fueron diversas, tratando de captar un amplio rango de operaciones reales en este proceso.
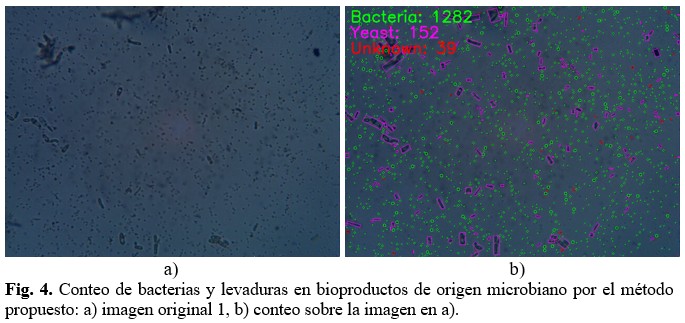
Como se puede apreciar en la figura 4, mediante el algoritmo desarrollado se logra detectar las bacterias y levaduras. En estas imágenes las bacterias fueron enmarcadas en color verde y las levaduras en color malva. Los microorganismos que hasta ahora se clasifican como desconocidos se enmarcan en rojo. (Fig. 4).
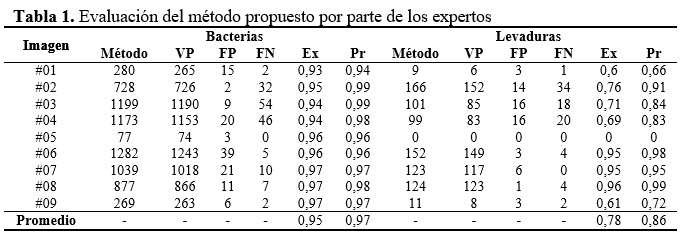
En la tabla 1 se pueden apreciar los resultados luego de ser evaluadas nueve imágenes por expertos en microbiología, así como el desempeño del método en cuanto a exactitud (Ex) y precisión (Pr) utilizando la Ecuación 2 y la Ecuación 3 respectivamente:
Es de mencionar que los casos que el método detectó correctamente, sean bacterias o levaduras, fueron clasificadas como verdaderos positivos (VP), los casos clasificados incorrectamente fueron contados como falsos positivos (FP), las bacterias o levaduras que el método no detectó fueron considerados como falsos negativos (FN), y no se hizo un conteo de verdaderos negativos (VN) pues la intención del método no es detectar aquellas siluetas en la imagen que no son microrganismos. Los contornos detectados como desconocidos no fueron tomados en cuenta en este análisis pues debe llevar un análisis por parte de los expertos para su posible clasificación. (Tabla 1).
Como se puede apreciar en los resultados al procesar las imágenes, la detección y conteo de bacterias se logra con una exactitud y precisión aceptable, en ambos casos por encima de 0,95; no en el caso de las levaduras cuya exactitud y precisión es considerablemente menor, alrededor de 0,78 la exactitud y 0,86 la precisión. En este caso el usuario debe discernir en aquellos contornos marcados como unknown cuál es su posible clasificación, o si debiera descartarlo.
Este hecho se encuentra estrechamente vinculado con la forma regular propia de las bacterias, en comparación con la forma menos regular proyectan las levaduras en las imágenes tomadas a estos compuestos.
Como es apreciable también en las imágenes hay sombras, artefactos que pertenecen a microorganismos que se encuentran por debajo de la capa de análisis o que están mal enfocados, lo cual provoca en los casos más claros que se clasifiquen como bacterias pues aparece por lo menos un contorno que cumple con todos los criterios de selección, y en la mayoría de los casos ese contorno no logra aparecer visiblemente claro.
También existen contornos y siluetas que no cumplen con los criterios de ajustes establecidos.
DISCUSIÓN
Como resultado de la amplia gama de condiciones experimentales reflejadas se detectaron diferentes escenarios donde el algoritmo puede tener un desempeño que no sea el óptimo.
Una de las condiciones que puede provocar errores de clasificación es la forma misma de los contornos en la imagen, especialmente las más grandes que generalmente se corresponden con las levaduras.
El simple hecho de poseer características de forma no homogéneas en las levaduras puede resultar en que, durante el procesamiento de las imágenes, parte de su forma sea removida de la imagen por las diferentes operaciones, resultando en una partición del mismo microorganismo en varias regiones de clasificación, como se puede observar en la figura 5-a.
A esto se le adiciona que en ocasiones la silueta del microorganismo en la imagen no se puede observar de manera completa, o que existen agrupamientos de bacterias y levaduras lo cual dificulta el proceso de clasificación y conteo, tal como se puede observar en la Figura 5-b y Figura 5-c donde la clasificación de levaduras (rectángulo malva) se encuentra compuestos por diferentes microorganismos. (Fig.5).
Otras problemáticas en contra de la obtención de buenos resultados ya han sido reportadas en investigaciones similares como suele ser el bajo contraste entre los microrganismos y la suspensión. Esta situación provoca que los métodos y algoritmos no puedan definir bien entre lo que es fondo y lo que debe ser detectado.
Existen también problemas asociados con el lente de la cámara o en donde se prepara la disolución (portaobjetos y del cubre objetos), que en ocasiones pueden contener suciedad u otros cuerpos que no sean los analizados en la investigación.
Otra de las dificultades a la hora de obtener buenos resultados con el algoritmo es el grado de enfoque con que es capturada la imagen, condición que puede ser perjudicial para cualquier algoritmo de visión por computador. En este caso el enfoque del conjunto microscopio-cámara sobre la muestra influye mucho en los resultados que se pueden obtener.
Alternativa de conteo con el software ImageJ.
El software ImageJ mencionado anteriormente también fue empleado para procesar imágenes de los bioproductos microbianos en estudio, y se observó cual era el desempeño del mismo.
El software ImageJ, al ser una herramienta muy completa, posibilita la obtención de los resultados del conteo en diferentes formas, además de que puede mostrar un resumen del conteo para un análisis más detallado por parte de los especialistas. Una de las opciones de visualizar los resultados es el empleo de la opción Overlay Masks, que posibilita ver qué contorno fue contado y le asigna un color a cada uno, tal como se puede apreciar en la figura 6. (Fig. 6).
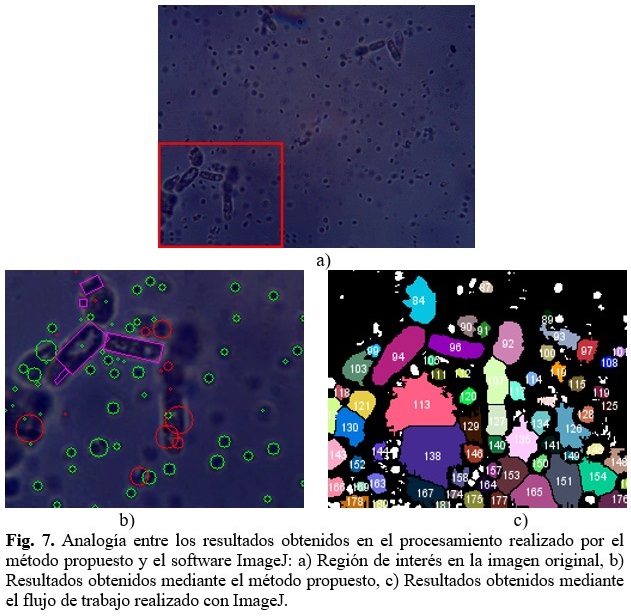
Si bien las formas pequeñas son detectadas correctamente con esta variante de procesamiento, en los contornos de mayor área se producen segmentaciones de las imágenes, y por ende clasificaciones y conteos que no son correctos, tal como se puede observar en la figura 7. (Fig.7).
El flujo empleado con el software ImageJ, al igual que el método propuesto, no logran establecer una clasificación exacta en el caso de los contornos grandes, es decir, las levaduras.
El método propuesto puede tender a hacer, debido al propio procesamiento de la imagen, una subclasificación de los contornos de las levaduras provocando la aparición de contornos marcados como desconocidos o falsos positivos de levaduras. En cambio, el método seguido con el software ImageJ puede provocar una sobreclasificación de los contornos analizados como levaduras, provocando de esta manera la aparición de muchos falsos positivos en la clasificación de las levaduras.
Se comprobó en los experimentos llevados a cabo que estos errores aumentan a medida que la magnificación aumenta, pues las áreas de los microorganismos, especialmente las levaduras, y las áreas no homogéneas del fondo, crecen y empiezan a generar falsos positivos.
Al igual que el método propuesto, los resultados obtenidos por el procesamiento realizado con ImageJ se ven afectado por las mismas situaciones que se describieron anteriormente y que han sido reportadas en la literatura.
La idea en trabajos futuros no es prescindir del software ImageJ, sino utilizarlo en provecho de la investigación dada las potencialidades del mismo, creando un método robusto para la detección de bacterias y levaduras mediante la combinación del método expuesto aquí y las herramientas propias del software.
Se pude concluir que, según se refiere en la literatura especializada y se comprobó durante el desarrollo de la presente investigación, es difícil la creación de métodos generales para la clasificación de microorganismos en imágenes digitales. Es por ello que un alto por ciento de la bibliografía actual en este tema se centre en contar microrganismos de una sola especie, siendo las bacterias las que más esfuerzos han recibido en ese sentido por parte de la comunidad científica.
El método propuesto en este trabajo, basándose en operaciones morfológicas, detección de contornos y análisis estadístico, logra clasificar y contar con elevada precisión y exactitud la presencia de bacterias en imágenes digitales. En el caso de las levaduras el desempeño del método propuesto fue algo inferior.
Los resultados alcanzados por el método se ven influenciados grandemente por la diversidad de tamaños y formas de los microrganismos, el grado de agrupamiento de los mismos, el contraste entre los microrganismos y el fondo, y las condiciones en que son adquiridas las imágenes.
Al emplear el software ImageJ para cumplir con el mismo objetivo de contar bacterias y levaduras se puede apreciar que los desempeños entre los métodos son similares, obteniéndose los mayores errores de clasificación y conteo en los contornos más largos, los cuales generalmente pertenecen a las levaduras.
Agradecimientos
A los autores les gustaría agradecer al personal del Laboratorio de Microbiología Aplicada del Instituto de Biotecnología de las Plantas por el esfuerzo y el tiempo dedicado en la realización de los experimentos.
Conflictos de interés
Los autores declaran que no tiene ningún conflicto de interés.
Contribuciones de los autores
Conceptualización: Jorge Peña Martín, Yelenys Alvarado-Capó.
Análisis formal: Jorge Peña Martín, Yelenys Alvarado-Capó, Rubén Orozco Morales, Tatiana Pichardo, Ailet Abreu López.
Investigación: Jorge Peña Martín.
Metodología: Jorge Peña Martín, Yelenys Alvarado-Capó, Rubén Orozco Morales, Tatiana Pichardo, Ailet Abreu López.
Recursos: Jorge Peña Martín, Yelenys Alvarado-Capó, Tatiana Pichardo.
Software: Jorge Peña Martín.
Supervisión: Jorge Peña Martín, Yelenys Alvarado-Capó, Rubén Orozco Morales, Tatiana Pichardo, Ailet Abreu López.
Validación: Jorge Peña Martín, Yelenys Alvarado-Capó, Tatiana Pichardo.
Visualización: Jorge Peña Martín.
Redacción- borrador original: Jorge Peña Martín.
Redacción-revisión y edición: Jorge Peña Martín, Yelenys Alvarado-Capó, Rubén Orozco Morales, Tatiana Pichardo, Ailet Abreu López.
Financiamiento
Universidad Central Marta Abreu de Las Villas. Villa Clara. Cuba.